检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
摘要
胚胎着床是包括人类在内的哺乳动物发育过程中的里程碑事件,生理状态下超过半数以上的人类胚胎由于无法顺利着床导致不孕。既往研究通常使用小鼠和食蟹猴等模式生物对这一过程展开探索,然而调控围着床时
期胚胎发育的分子机制和形态学变化特征在不同物种之间存在较大差异,这使得在小鼠等模式生物研究中获得的调控规律较难为人类胚胎发育研究提供有价值的线索。然而,由于人类胚胎着床发生在受精卵形成后一周左右的时间点,这使得研究者们无法获得生理状况下的这一发育阶段的人类胚胎。长期以来,这一人类关键发育阶段一直成为发育生物学研究的黑匣子。为了深入探讨这一过程中的分子动态规律,挖掘调控胚胎着床过程中的潜在分子机制,2019年8月22日,永利官网、北京未来基因诊断高精尖创新中心(ICG)、生物医学前沿创新中心(BIOPIC)汤富酬课题组携手北京大学第三医院乔杰课题组合作在Nature在线发表了题为Reconstituting the transcriptome and DNA methylome landscapes of human implantation的研究论文。结合体外模拟人类着床策略1和高精度单细胞多组学测序技术2,3(single-cell RNA-seq, single-cell Trio-seq2),首次利用单细胞转录组和DNA甲基化组图谱重构了人类胚胎着床过程,系统解析了这一关键发育过程中的基因表达调控网络和DNA甲基化动态变化过程。研究者们发现围着床时期人类胚胎的三个主要细胞谱系(上胚层、原始内胚层、滋养外胚层)表现出独特的发育特征,而且胚胎在这一时期迅速呈现出母胎连接预备状态;随后,作者发现在可观察事件窗口期内(体外培养day12以前),雌雄胚胎的X染色体剂量并未达到平衡,且雌性胚胎逐渐启动并逐渐呈现出父源或母源X染色体随机失活趋势;最后,研究者们利用单细胞多组学测序手段发现不同细胞谱系具有截然不同的DNA甲基化动态变化特征,提示基因表达调控网络和DNA甲基化可能共同协调决定囊胚阶段后的细胞谱系命运决定。
引言
人类胚胎发育从具有全能性的受精卵开始,伴随着多次卵裂和分裂从而形成一个具有特定结构的囊胚。囊胚主要由外围的外滋养层细胞(trophectoderm,TE)和内细胞团(inner cell mass,ICM)细胞组成,而成熟的内细胞团会进一步发育成为具有多能性的上胚层(epiblast,EPI)和原始内胚层(primitive endoderm,PE)。囊胚着床之后TE主要分化为胚外滋养层结构,例如胎盘等;而上胚层主要会发育形成胎体的各个组织器官。随着体外受精技术的革新,人类早期着床前胚胎发育过程被广泛研究 ADDIN EN.CITE ADDIN EN.CITE.DATA 4,5。然而,在发育的第5-7天,胚胎必须通过着床进入母体子宫壁才能继续存活和发育,随后逐渐形成原肠胚,并进一步分化形成各类器官和组织原基。
着床失败是导致早期流产的重要因素之一6。然而研究者们几乎无法获得自然受孕后的早期着床后阶段的人类胚胎,导致人们对胚胎发育过程中这一关键阶段的谱系特化和分子调控等规律知之甚少。猴子胚胎曾被用于早期胚胎发育研究7,但着床后早期胚胎的猴子模型与人类仍然存在一定的物种差异。小鼠作为最常见模式生物被广泛应用于早期胚胎研究。人类囊胚与子宫内膜细胞共培养体系为模拟体内着床后发育过程提供了另一实验策略8,然而这类研究策略对于着床后发育过程的重现程度和可重复性存在一定问题。尤其是该体系必须有母体组织的参与,因此较难被推广使用,且无法应用于胚胎的自主性构建(self-organization)研究。2016年,Magdalena Zernicka-Goetz实验室建立了小鼠着床后胚胎的体外培养体系,该研究组利用独特的培养条件将小鼠着床前的囊胚进行体外培养,直至形成类似于着床后胚胎的三胚层结构,为阐明早期着床后胚胎发育提供了重要实验手段9。
然而,不同哺乳动物胚胎类型之间存在较明显的物种差异,说明从小鼠到人类之间的发育过程的研究推理从而获得的有效结论非常有限,这意味着人类着床后胚胎形态学和分子调控等变化规律最好能直接通过人类胚胎研究来获取。相比小鼠来说,人类囊胚形成过程中的分子和细胞学机制研究仍然偏少,已有研究暗示二者在谱系分化和功能方面存在时间和潜能上的巨大差异10。Ali H. Brivanlou实验室以及Magdalena Zernicka-Goetz实验室近期将小鼠着床后胚胎培养体系进一步发展至人类胚胎,新建的培养体系同样能促进人类胚胎在体外经历着床前向着床后的转化与发育,培养体系无需母体组织参与;形态学证据揭示了在这一阶段人类胚胎发育的主要特征,表明人类胚胎在着床后期具有自我构建(self-organization)特性,这极大的丰富了人们对于人类胚胎着床这一生物学过程的认识1,11。
细胞身份识别和命运取决于其基因表达网络,同时该网络受到复杂的遗传和表观遗传学等机制调控。解析人类胚胎细胞基因表达的时空特异性模式对于了解胚胎早期发育过程至关重要。将单细胞分辨率组学检测技术应用于人类胚胎发育研究使人们精确认识这一过程成为了可能。汤富酬课题组和乔杰课题组长期紧密合作,致力于包括着床前胚胎在内的人类生殖系细胞发育过程中基因表达、表观遗传学调控特征和潜在的机制研究:2013年,结合汤富酬课题组发展的单细胞转录组测序技术12,绘制了人类着床前胚胎的高精度单细胞基因表达特征图谱(Nature Structural & Molecular Biology,201313;2014年,该课题组利用新发展的微量细胞DNA甲基化组测序技术系统解析了人类着床前胚胎发育过程中DNA甲基化组重编程动态特征(Nature,201414;2018年,该团队利用单细胞DNA甲基化高通量测序技术在单细胞水平解析了人类着床前胚胎发育的DNA甲基化组图谱(Nature Genetics,201815。同年,他们利用单细胞多组学测序技术解析了人类着床前胚胎发育过程中DNA甲基化组和染色质状态组的重编程过程,以及染色质状态与DNA甲基化之间的相互关系(Nature Cell Biology,201816。以上各项研究极大丰富了人们对于人类着床前胚胎基因表达和表观遗传调控规律的认知。此外,多个课题组曾利用一系列单细胞技术对人类着床前胚胎的分子表达谱进行了系统性解析 17,18。然而受困于技术限制,胚胎着床这一关键发育阶段的多维度分子表达特征,尤其是该过程中谱系特异性分子表达谱和潜在的调控规律长期以来一直是未解之谜。
为此,两个课题组再次携手合作,结合人类胚胎体外培养体系与单细胞组学技术(单细胞转录组和多组学测序),系统揭示了人类早期着床后胚胎发育过程中的分子表达图谱,并在此基础上挖掘了基因表达调控网络、DNA甲基化动态特征等潜在分子调控机制。
结果
1. 人类胚胎在囊胚阶段后期具备体外自我生长能力(体外模拟胚胎着床)
为了证实胚胎在体外无母体组织参与情况下的着床过程,研究者根据已报道胚胎培养策略重现了胚胎形态学动态变化特征 ADDIN EN.CITE ADDIN EN.CITE.DATA 1,11。证据显示胚胎在无母体组织参与下能发育至第12/14天,胚胎逐渐呈现出特异性的形态学特征,例如滋养外胚层细胞数量大幅度增加,上胚层逐渐形成羊膜腔以及原始内胚层逐渐生长并逐渐包围上胚层(图1,视频1)。根据国际伦理学准则,研究者们在第14天终止了体外胚胎培养实验。
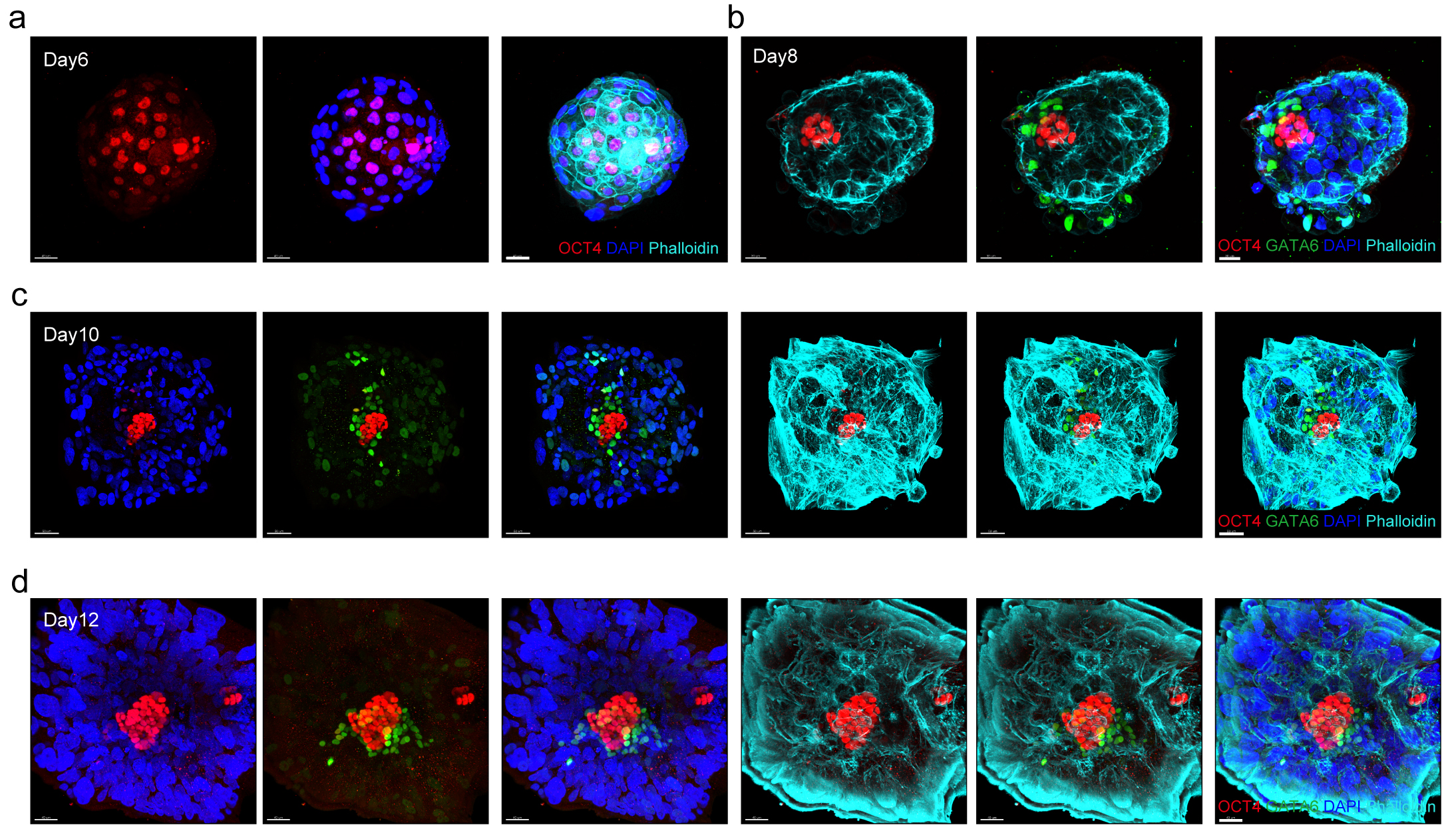
图1 人类胚胎体外模拟着床生长过程(day6-12)
您的浏览器不支持 video 标签。
视频1 人类围着床时期胚胎三维重建结构(day12)
2. 不同细胞谱系的关键发育特征以及特征性基因挖掘
研究者利用已知的谱系标志基因对围着床时期的胚胎细胞进行了谱系鉴定,结果显示胚胎基本维持了囊胚晚期的三个主要细胞谱系(上胚层、原始内胚层、滋养外胚层)。有趣的是,各个谱系均逐渐呈现出各自独特的基因表达特征,例如上胚层呈现出明确的多能性转变(pluripotency transition),原始内胚层则逐渐开始表达卵黄囊发育相关基因(例如CD44),而滋养外胚层则逐渐开始表达荷尔蒙相关基因(例如CGB家族基因),以上特征均提示胚胎在这一关键发育阶段逐渐呈现出母胎连接预备状态(图2)。不同细胞谱系之间的基因表达对比分析,各类细胞谱系均具备独特的特征性基因(signature gene)。且部分基因与已发表食蟹猴围着床时期胚胎相关谱系的基因表达特征类似(图3)。
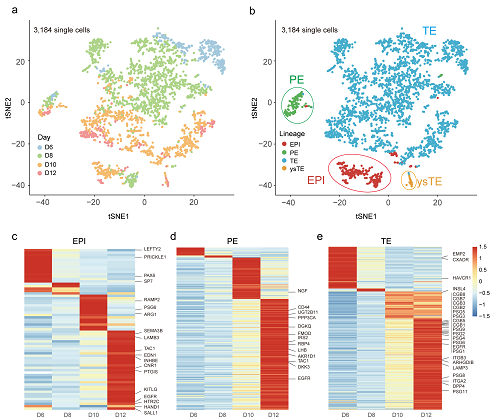
图2 人类围着床时期胚胎的谱系鉴定和各自的发育学特征
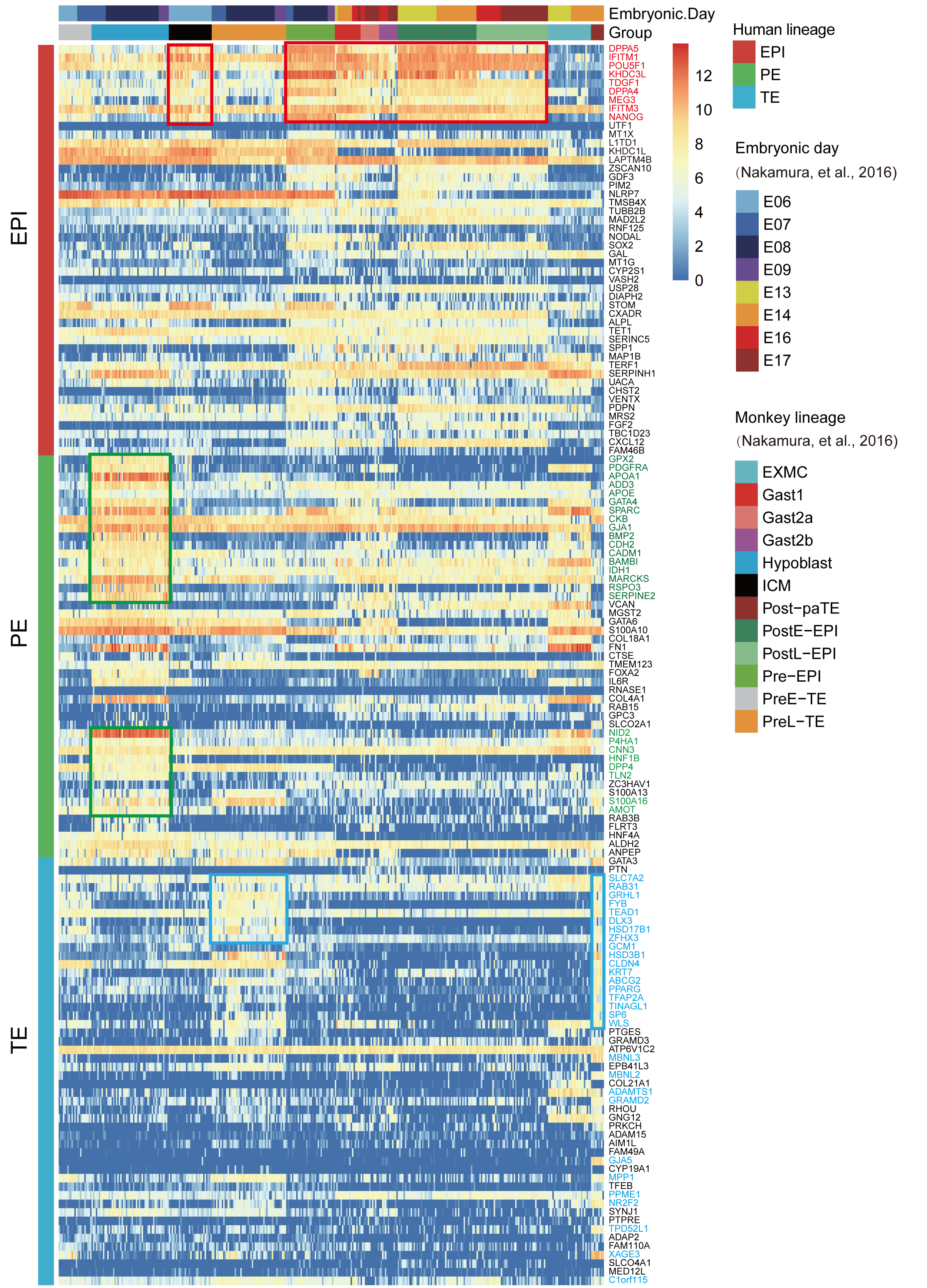
图3人类与食蟹猴着床后胚胎细胞的基因表达图谱对比分析
另一方面,OTX2过去曾被报道是围着床时期胚胎上胚层(EPI)多能性转化(pluripotency transition)的重要标志基因19,然而在本研究中的转录组数据显示OTX2主要表达在原始内胚层(PE),而并非上胚层(EPI)。进一步全胚胎免疫荧光染色显示OTX2表达在部分原始内胚层细胞中,与转录组分析结果一致。这表明具有相同发育来源(内细胞团起源)的两类细胞(上胚层细胞和原始内胚层细胞)可能携带特殊基因表达痕迹(图4)。这些数据提示该研究中转录组数据的潜在资源价值,新的谱系标记基因鉴定可能有助于早期胚胎中的谱系鉴定和干/祖细胞衍生研究。
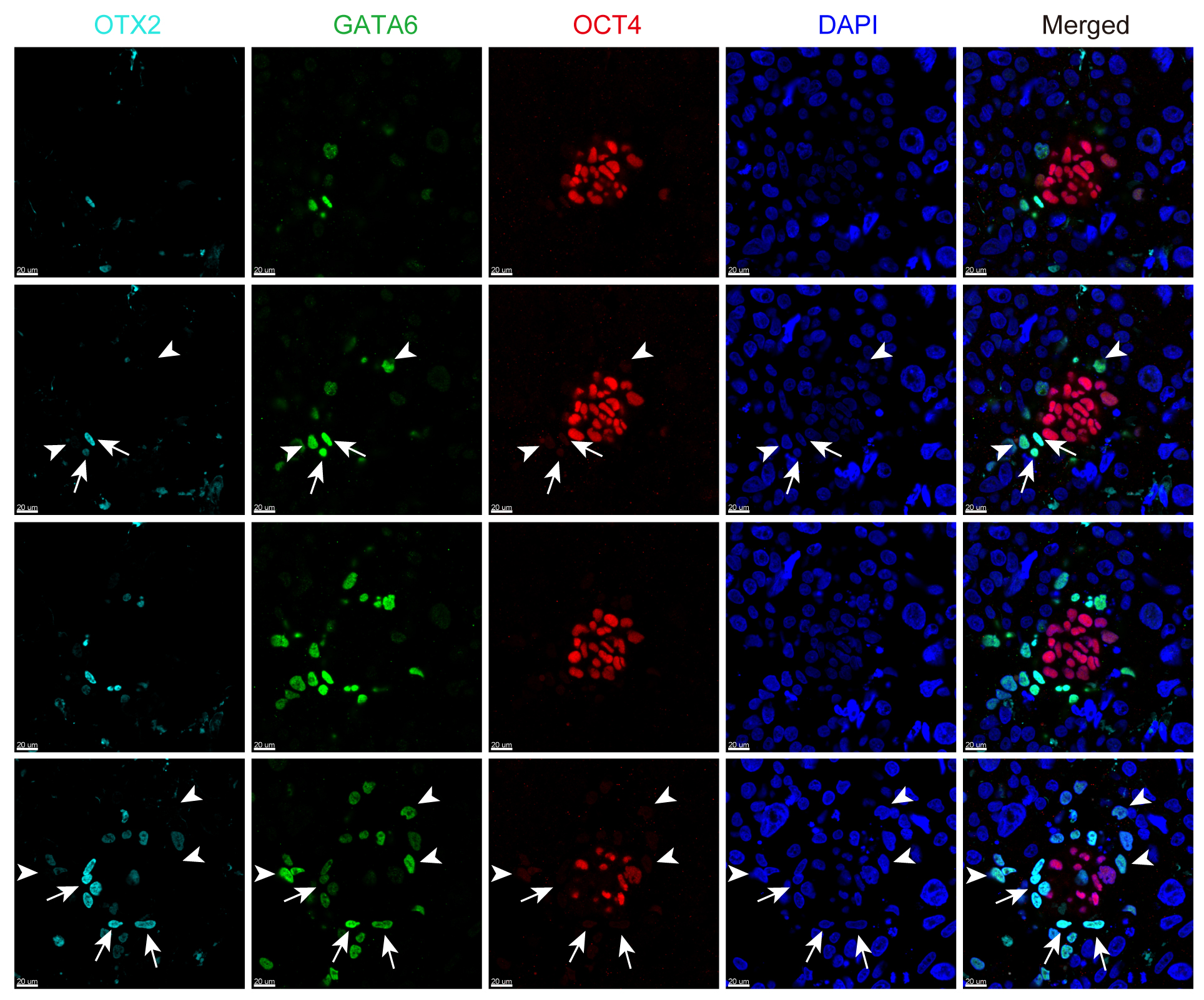
图4 OTX2在围着床时期胚胎的表达情况
3. 滋养外胚层特化成为两类亚群
研究者们发现随着围着床时期胚胎的发育,滋养外胚层逐渐形成两类亚群,其中一类亚群主要表达女性妊娠相关基因,而另一类亚群则几乎不表达该类基因。随后这两类细胞被鉴定为细胞滋养层细胞(cytotrophoblasts,CTs)和合胞滋养层细胞(syncytiotrophoblasts,STs),除激素相关基因外,合胞滋养层细胞(STs)还表达部分新发现的基因,例如TCL6和TBX3(图5)。
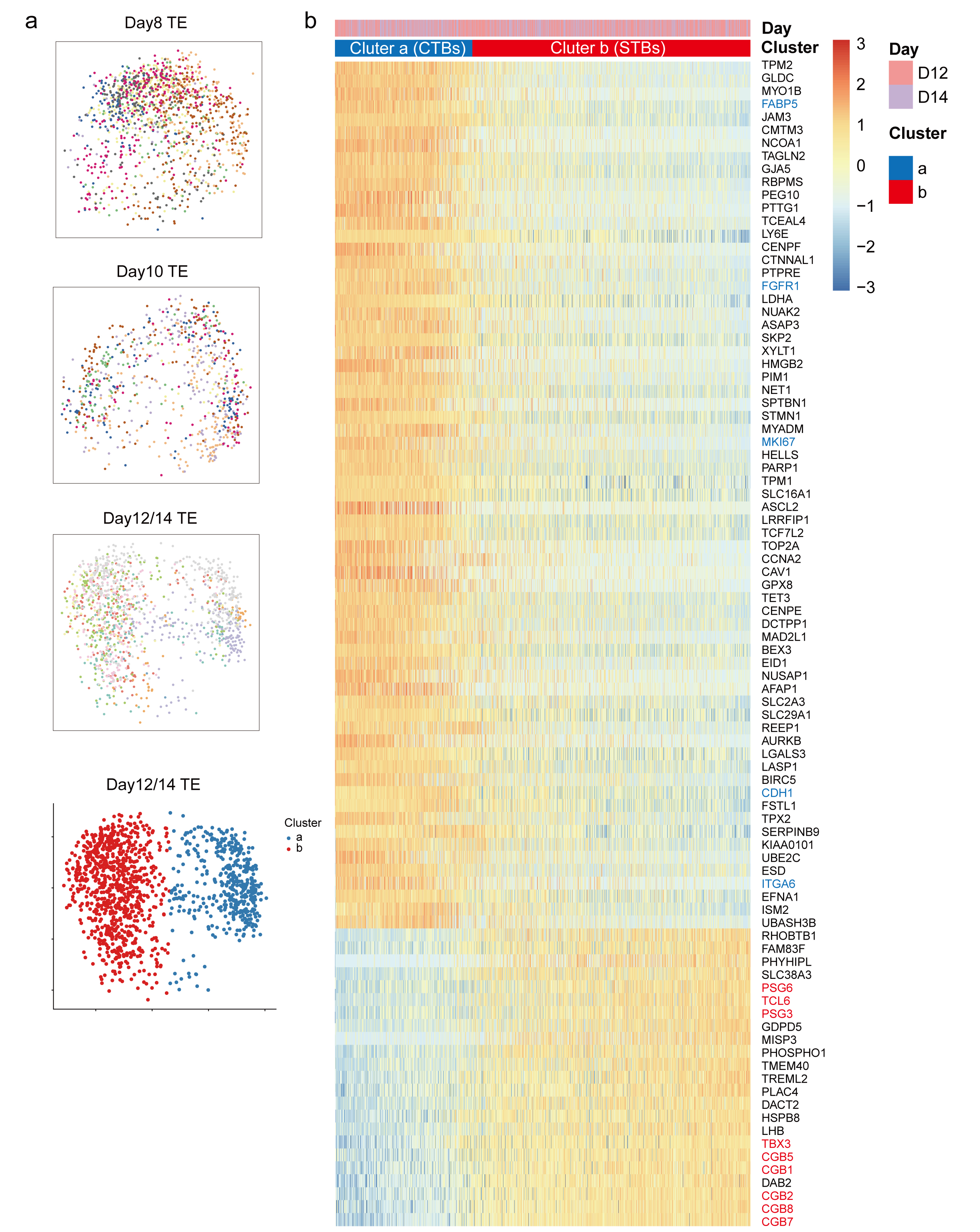
图5 滋养外胚层(TE)两类亚群的特征性基因表达谱
4. X染色体剂量平衡
自1961年Lyon等人报道雌雄个体之间的X染色体剂量特征后,X染色体剂量平衡一直是发育生物学关注的焦点之一20。X染色体失活(XCI)对于女性(XX)与男性(XY)之间X连锁基因的剂量平衡具有重要意义,同时X染色体上调(XCU)对于X连锁基因与常染色体基因之间的剂量平衡具有重要意义。2016年Fredrik Lanner团队曾报道雌雄胚胎的X染色体剂量在囊胚阶段几乎已达平衡,且为X染色体阻滞模式(X-dampening,雌性细胞两条X染色体剂量均部分下调)17。在本研究中,作者们捕获到了多个阶段的雌雄胚胎,这为系统揭示这一过程中的关键生物学细节提供了可能。可追踪父母源等位基因表达的单细胞全长转录组测序分析显示,在可观察事件窗口内(day12以前),雌雄胚胎的X染色体剂量并未达到平衡,且雌性胚胎逐渐启动并逐渐呈现出父源或者母源X染色体随机失活趋势(XCI)(图6)。另一方面,X染色体剂量应与常染色体基因的表达量平衡则需要雌性与雄性中的X染色体上调(XCU)来实现,在晚期胚胎单细胞中活跃的那条X染色体(雄性细胞中仅含一条X染色体,处于活跃状态;雌性细胞则含有一条活跃的X染色体和一条失活的X染色体)需要上调表达剂量两倍,达到跟同一个细胞中每个常染色体两个拷贝同样的表达剂量(X染色体/常染色体的表达剂量比从1:2上调到2:2)。该研究发现X染色体上调在着床阶段的雌性和雄性胚胎细胞中均已经启动,但是还没有达到上调两倍表达剂量的完成状态。此外,研究者们还发现围着床时期胚胎还存在着一定程度的拷贝数变异(CNV),且拷贝数变异并未影响主要谱系的整体基因表达特征。
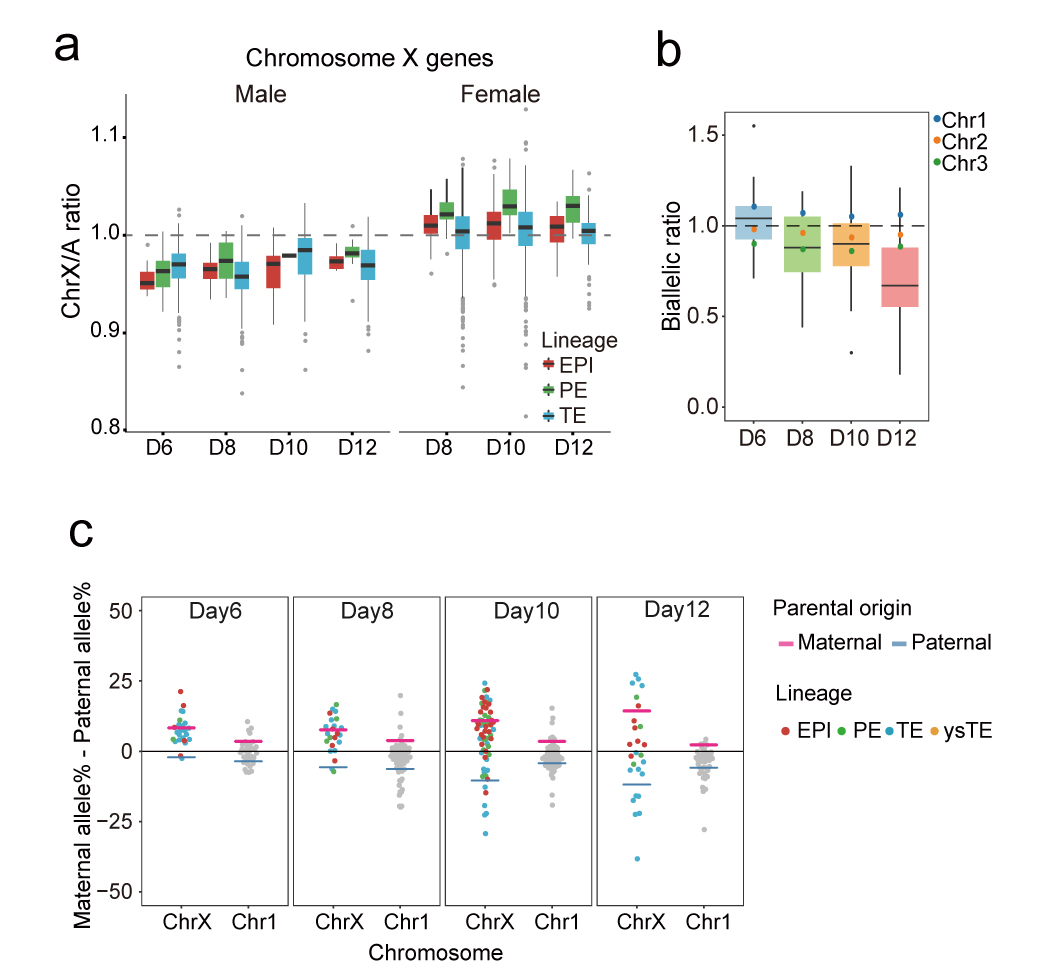
图6 围着床时期胚胎的X染色体剂量动态变化过程
5. 谱系特异性DNA甲基化动态变化规律
为进一步分析胚胎着床过程中的基因组甲基化特征,研究者们利用该团队创建的单细胞多组学测序技术对三类细胞谱系的基因组甲基化过程进行了深度分析。围着床阶段胚胎细胞在DNA甲基化特征水平分布成4个主要细胞群体(图7):囊胚期(day 6)上胚层/原始内胚层/滋养外胚层、着床后时期(day8/10)上胚层、着床后时期(day8-12)原始内胚层、以及着床后时期(day8-12)滋养外胚层。该结果说明三类细胞谱系在囊胚发育阶段(着床前)具有相似的DNA甲基化模式,在着床后迅速获得了各自独特的DNA甲基化特征。
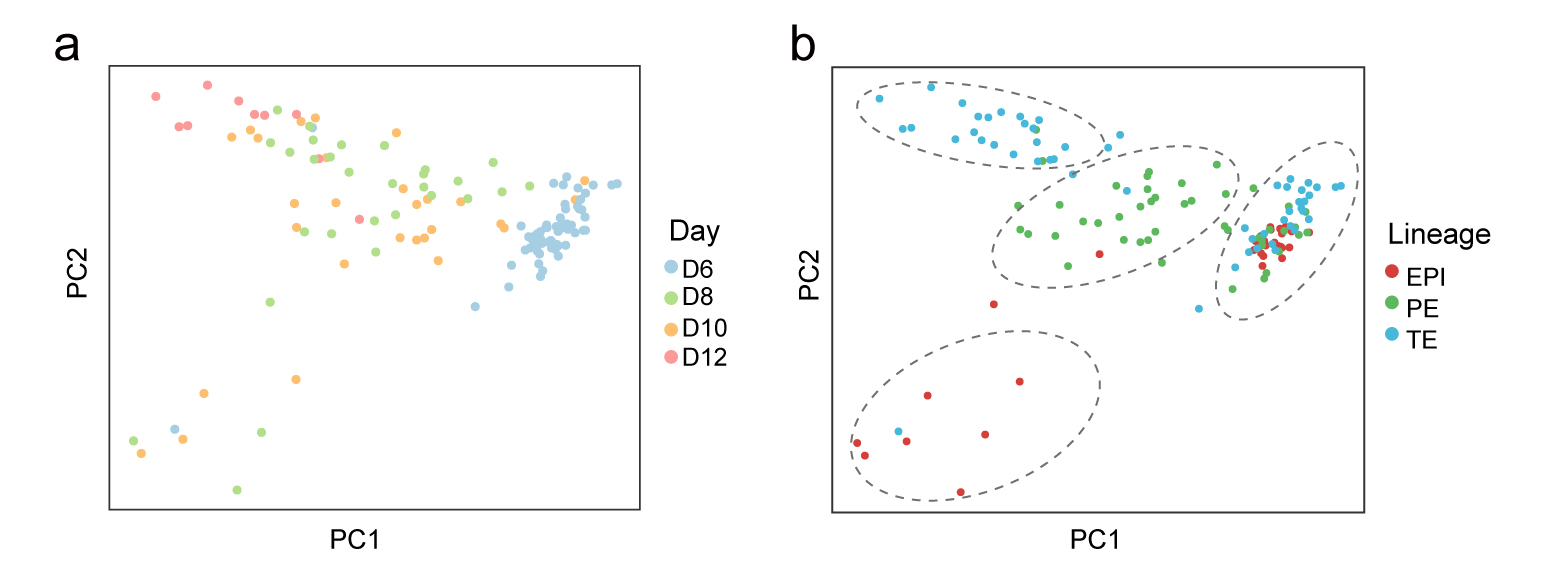
图7 各个细胞谱系在着床过程中的具有特异性DNA甲基化特征(基于PCA分析)
接下来,研究者重点关注三个细胞谱系各自的DNA甲基化动力学变化过程。总体来说,上胚层、原始内胚层和滋养外胚层在囊胚发育阶段后基因组均发生了重新甲基化(图8),表明胚胎在着床过程中可能经历了表观遗传调控的自我构建过程。其中上胚层的DNA甲基化水平从囊胚的26.1%(day6)显著增加到着床后胚胎的60.0%(day10)。同样, 滋养外胚层群体的DNA甲基化水平也在囊胚阶段(day6)从23.5%上升到着床后胚胎(day10)的46.3%。滋养外胚层群体的DNA甲基化水平显著低于上胚层谱系(day10),这意味着胚内(上胚层)和胚外(滋养外胚层)谱系细胞的表观遗传调控过程可能存在较大差异。出乎意料的是,囊胚期原始内胚层的DNA甲基化水平(27.0%)与上胚层相当(26.1%),第8天上升至33.2%(上胚层为49.9%),第10天略有上升至36.8% (上胚层为60.0%)。与上胚层相比,来自内细胞团的另一个谱系原始内胚层在着床过程中意外地呈现出非常缓慢的DNA甲基化动力学特征(图8)。这些数据表明,着床后早期胚胎在囊胚期后很快起始了大规模DNA甲基化过程,三个主要谱系不仅表现出不同的基因表达特征,而且在DNA重甲基化特征呈现不同步的明显差异。这表明DNA甲基化可能在维持特定细胞谱系的发育过程中发挥重要作用。因此,这也暗示在着床过程中的调控各个细胞谱系发育的表观遗传学特征也存在差异。
有趣的是,不同谱系间差异DNA甲基化基因分析表明,各个谱系在胚胎第10天都携带特异性DNA甲基化。例如,多能性基因POU5F1和NANOG在第8天滋养外胚层细胞中被特异性甲基化,而上胚层和原始内胚层细胞则没有。相反,滋养外胚层发育基因如MMP26和PSG7在上胚层和原始内胚层细胞中特异性甲基化,但在第8天滋养外胚层细胞中却维持着低甲基化的状态 ADDIN EN.CITE ADDIN EN.CITE.DATA 21,22。相比之下,在第8天,原始内胚层标记基因如APOA1和CPN1在上胚层和滋养外胚层细胞中特异性甲基化,而在原始内胚层细胞中没有被甲基化(图7)23,24。这些结果表明,DNA甲基化在着床后早期的时间窗口内可能参与特异性调控了关键基因的转录,与基因转录共同协调决定了不同细胞谱系的发育潜能特化过程。
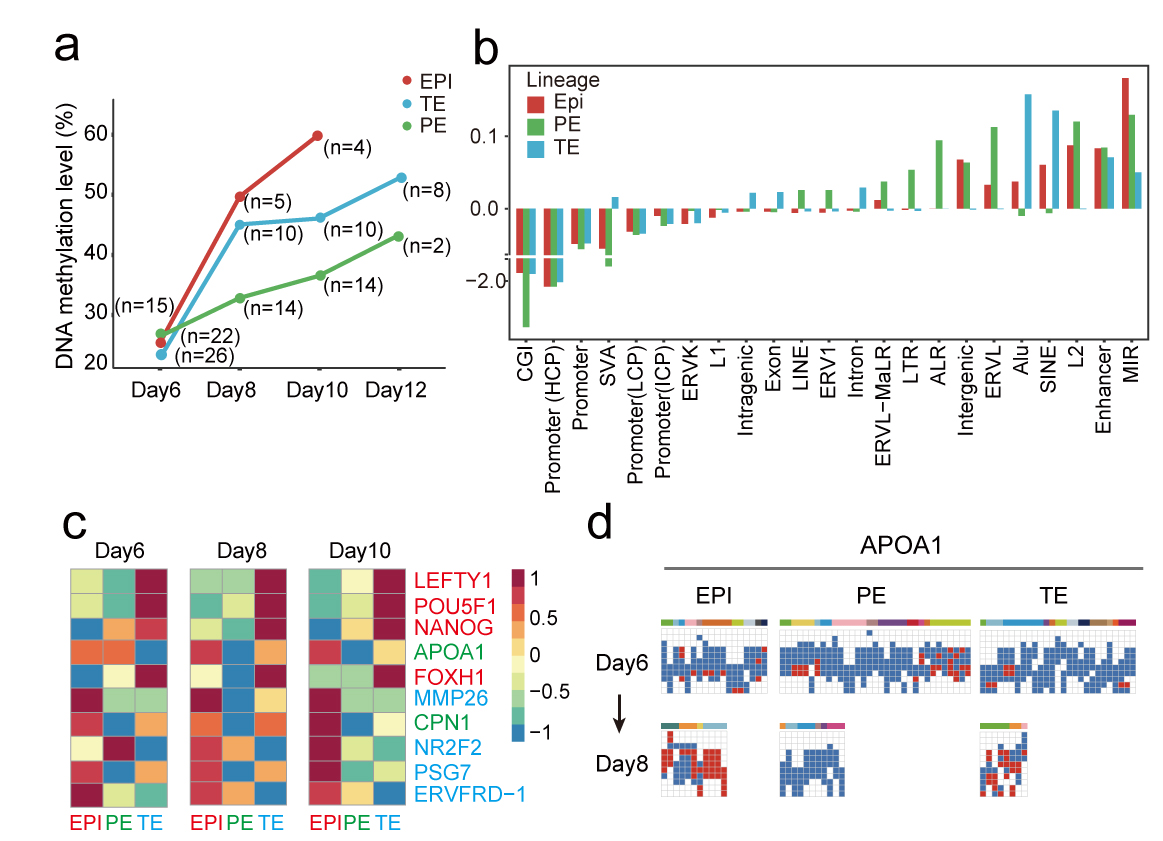
图8 三类主要谱系在围着床发育时期的DNA甲基化水平动态变化过程
讨论
综上,本研究提供的单细胞分辨率的转录组和DNA甲基化组数据对于研究早期人类胚胎发育研究具有潜在的重要价值。此外,尽管仍不清楚体内和体外胚胎发育之间存在着的具体差异,但本研究提供的数据资源可能为优化模拟体外着床策略提供直接线索与帮助,这也为多能干细胞的分化与发育相关研究提供重要线索和依据。
永利官网博士后周帆博士、博士生汪睿、北京大学第三医院博士生袁鹏、博士生任一昕和永利官网博士生毛雨诺为该论文的并列第一作者,北京大学北京未来基因诊断高精尖创新中心/生物医学前沿创新中心汤富酬教授和北京大学第三医院乔杰教授为论文的共同通讯作者。该课题得到了北京大学生命科学仪器中心(成像平台)和北京大学高精尖中心高通量测序平台的协助与支持。
原文链接:https://www.nature.com/articles/s41586-019-1500-0
参考文献
1 Deglincerti, A. et al. Self-organization of the in vitro attached human embryo. Nature 533, 251-254, doi:10.1038/nature17948 (2016).
2 Li, L. et al. Single-Cell RNA-Seq Analysis Maps Development of Human Germline Cells and Gonadal Niche Interactions. Cell stem cell 20, 858-873 e854, doi:10.1016/j.stem.2017.03.007 (2017).
3 Bian, S. et al. Single-cell multiomics sequencing and analyses of human colorectal cancer. Science 362, 1060-1063, doi:10.1126/science.aao3791 (2018).
4 Edwards, R. G., Bavister, B. D. & Steptoe, P. C. Early stages of fertilization in vitro of human oocytes matured in vitro. Nature 221, 632-635 (1969).
5 Edwards, R. G., Steptoe, P. C. & Purdy, J. M. Fertilization and cleavage in vitro of preovulator human oocytes. Nature 227, 1307-1309 (1970).
6 Koot, Y. E., Teklenburg, G., Salker, M. S., Brosens, J. J. & Macklon, N. S. Molecular aspects of implantation failure. Biochimica et biophysica acta 1822, 1943-1950, doi:10.1016/j.bbadis.2012.05.017 (2012).
7 Nakamura, T. et al. A developmental coordinate of pluripotency among mice, monkeys and humans. Nature 537, 57-62, doi:10.1038/nature19096 (2016).
8 Weimar, C. H., Post Uiterweer, E. D., Teklenburg, G., Heijnen, C. J. & Macklon, N. S. In-vitro model systems for the study of human embryo-endometrium interactions. Reproductive biomedicine online 27, 461-476, doi:10.1016/j.rbmo.2013.08.002 (2013).
9 Bedzhov, I., Leung, C. Y., Bialecka, M. & Zernicka-Goetz, M. In vitro culture of mouse blastocysts beyond the implantation stages. Nature protocols 9, 2732-2739, doi:10.1038/nprot.2014.186 (2014).
10 Rossant, J. Mouse and human blastocyst-derived stem cells: vive les differences. Development 142, 9-12, doi:10.1242/dev.115451 (2015).
11 Shahbazi, M. N. et al. Self-organization of the human embryo in the absence of maternal tissues. Nature cell biology 18, 700-708, doi:10.1038/ncb3347 (2016).
12 Tang, F. et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature methods 6, 377-382, doi:10.1038/nmeth.1315 (2009).
13 Yan, L. et al. Single-cell RNA-Seq profiling of human preimplantation embryos and embryonic stem cells. Nature structural & molecular biology 20, 1131-1139, doi:10.1038/nsmb.2660 (2013).
14 Guo, H. et al. The DNA methylation landscape of human early embryos. Nature 511, 606-610, doi:10.1038/nature13544 (2014).
15 Zhu, P. et al. Single-cell DNA methylome sequencing of human preimplantation embryos. Nature genetics 50, 12-19, doi:10.1038/s41588-017-0007-6 (2018).
16 Li, L. et al. Single-cell multi-omics sequencing of human early embryos. Nature cell biology 20, 847-858, doi:10.1038/s41556-018-0123-2 (2018).
17 Petropoulos, S. et al. Single-Cell RNA-Seq Reveals Lineage and X Chromosome Dynamics in Human Preimplantation Embryos. Cell 167, 285, doi:10.1016/j.cell.2016.08.009 (2016).
18 Wu, J. et al. Chromatin analysis in human early development reveals epigenetic transition during ZGA. Nature 557, 256-260, doi:10.1038/s41586-018-0080-8 (2018).
19 Shahbazi, M. N. et al. Pluripotent state transitions coordinate morphogenesis in mouse and human embryos. Nature 552, 239-243, doi:10.1038/nature24675 (2017).
20 Lyon, M. F. Gene action in the X-chromosome of the mouse (Mus musculus L.). Nature 190, 372-373 (1961).
21 Qiu, W. et al. Spatio-temporal expression of matrix metalloproteinase-26 in human placental trophoblasts and fetal red cells during normal placentation. Biology of reproduction 72, 954-959, doi:10.1095/biolreprod.104.036772 (2005).
22 Camolotto, S. et al. Expression and transcriptional regulation of individual pregnancy-specific glycoprotein genes in differentiating trophoblast cells. Placenta 31, 312-319, doi:10.1016/j.placenta.2010.01.004 (2010).
23 Yadgary, L., Wong, E. A. & Uni, Z. Temporal transcriptome analysis of the chicken embryo yolk sac. BMC genomics 15, 690, doi:10.1186/1471-2164-15-690 (2014).
24 Gerovska, D. & Arauzo-Bravo, M. J. Does mouse embryo primordial germ cell activation start before implantation as suggested by single-cell transcriptomics dynamics? Molecular human reproduction 22, 208-225, doi:10.1093/molehr/gav072 (2016).