检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2017年5月9日国际专业基因组学研究刊物Genome Research在线发表了北京大学白凡、谢晓亮团队与天津医科大学张宁团队合作研究成果。作者团队使用单细胞测序技术对来自同一病人的循环肿瘤细胞和单个原发灶癌细胞的拷贝数变异进行分析,揭示了在肿瘤转移过程中基因拷贝数变异的演化过程。作者团队进一步对来自结直肠癌、乳腺癌、胃癌、前列腺癌多个癌种的病人外周血循环肿瘤细胞进行分析,研究了不同癌种的循环肿瘤细胞基因组特征与差异,具有重要的科学意义和临床应用价值。
癌症是世界范围内严重威胁人类健康的疾病。在中国,癌症的发病率、死亡率也一直居高不下。肿瘤的转移是导致癌症病人死亡的主要原因。早在1869年,澳大利亚籍医生Ashworth首次在癌症病人血液样品中观察到循环肿瘤细胞(CTC)的存在。在肿瘤转移过程中,癌细胞从原发肿瘤脱落,进入血液或淋巴循环系统,其中一些具有高度转移潜能的肿瘤细胞在循环系统中存活下来,成为循环肿瘤细胞,并进一步发展为远端器官转移肿瘤。循环肿瘤细胞是肿瘤发生远处转移的必经步骤,因此在外周血中检测到肿瘤细胞预示着有发生肿瘤转移的可能。对于循环肿瘤细胞的研究一直是癌症研究中的热点。
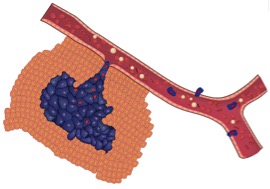
原发灶肿瘤细胞侵袭血管,形成循环肿瘤细胞
在本研究中,通过对一例结直肠癌病人原发灶肿瘤组织的单细胞,单个循环肿瘤细胞以及转移灶肿瘤组织基因组分析,作者团队发现原发灶肿瘤细胞的拷贝数变异经历不同的中间状态逐步积累,最终趋于在单个循环肿瘤细胞和转移灶组织观察到的相同的拷贝数变异模式。针对局部关键基因拷贝数变化的分析揭示,肿瘤转移伴随着抑癌基因PTEN的拷贝数逐步缺失及癌基因MYC的拷贝数逐步扩增。这个现象提示肿瘤的转移过程存在基因组拷贝数变化的趋同选择,同时有力地支持了肿瘤转移过程并非随机发生而是存在较强选择的假说。

结直肠癌病人原发灶肿瘤单细胞,循环肿瘤细胞和转移灶癌组织基因组拷贝数模式以及PTEN,MYC基因拷贝数变化
为了进一步阐述肿瘤细胞基因拷贝数异常形成的机制,作者团队对该病人癌基因MYC所在的8号染色体的4个拷贝数增加区域进行了单碱基水平的单细胞结构变异分析,并提出一个形成多区间拷贝数变异的两步模型: 首先在DNA复制过程中由于一系列的复制叉停顿和模板转位(FoSTeS)形成多区间拷贝数增加;随后通过同源重组(Homologous Recombination, HR)进一步将该区域扩增到较高的拷贝数,最终形成肿瘤细胞中异常的拷贝数模式。
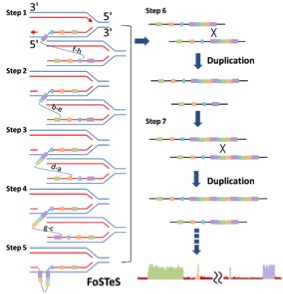
肿瘤细胞形成多区间拷贝数变异的机制模型
鉴于基因拷贝数变异在癌症发展与转移中的重要作用,作者团队对来自结直肠癌、乳腺癌、 胃癌、前列腺癌多个癌种的病人大量外周血循环肿瘤细胞进行分析。多癌种循环肿瘤细胞分析揭示同一个病人的不同CTC都展现出高度一致的全基因组拷贝数变化模式。这种现象在多个癌种的病人中都得到了验证,与作者团队早期在肺癌上的研究结果一致(Ni et al., PNAS, 2013)。但是在不同的癌种中,不同病人间CTC的CNA模式相似性存在较大的差异。例如在胃癌中,来自不同病人的CTC的CNA模式高度相似;而在乳腺癌中,来自不同病人的CTC的CNA模式差异明显。
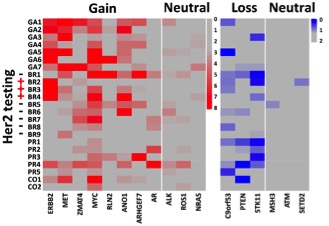
多癌种单循环肿瘤细胞中关键癌基因与抑癌基因变异情况
通过对单细胞测序数据的生物信息学分析,作者团队展示在多个癌种中可以从单个循环肿瘤细胞准确获取病人关键癌基因与抑癌基因的变异情况,如靶向治疗的靶点基因及导致耐药的基因变异等个体化治疗相关的重要信息。以CTCs检测为代表的癌症“液体活检”技术,可以避免反复穿刺给病人带来的伤害和痛苦, 能动态反映病人的疾病进程以及治疗过程中的疗效和预后,具有重大的临床应用价值。
北京大学永利官网生物动态光学成像中心白凡研究员、谢晓亮教授以及天津医科大学张宁教授为该论文的共同通讯作者。北大博士生高妍、苏哲、哈佛大学博士后倪晓晖博士、天津医科大学肿瘤医院郭华博士、巴一教授是这篇论文的并列第一作者。该项研究得到了科技部863计划,973计划,精准医学重大专项,国家杰出青年基金以及北京市科委的支持。
论文链接:“Single-cell sequencing deciphers a convergent evolution of copy number alterations from primary to circulating tumour cells”
http://genome.cshlp.org/content/early/2017/05/08/gr.216788.116.abstract